在细胞信号传导的复杂网络中,两个原本不直接相互作用的蛋白质如何通过中间“桥梁”蛋白实现功能联动,一直是领域内的研究难点。云南大学生命科学学院宁文秀团队在中科院一区nature index期刊《PLOS Biology》上发表了一项最新研究,开发出一种名为STUPPIT的新型邻近标记工具。该工具能够特异性标记并捕获充当“桥梁”的中间蛋白,为揭示信号通路中未知的蛋白质相互作用提供了创新的方法学解决方案。

Ø 信号通路中 “桥梁蛋白”的鉴定难题
解析信号通路的复杂性对于揭示组织发育、稳态维持及疾病发生机制至关重要。细胞信号通路依赖于精确调控的蛋白质-蛋白质相互作用,这些相互作用形成动态网络,调控基本生物学过程。传统的邻近标记技术,如TurboID、APEX2、PUP-IT等,能有效识别与特定“诱饵蛋白”直接相互作用的蛋白。升级版本的split-BioID、split-TurboID和split-APEX2则可识别两个已知直接相互作用蛋白周围的邻近蛋白。然而,对于两个存在功能关联但无直接接触的蛋白质,其间起连接作用的“桥梁蛋白”仍难以被直接捕获和鉴定。这类中间蛋白可能广泛存在于Hippo、Wnt、Hedgehog等重要信号通路中,是理解信号转导与通路间相互作用的关键。
Ø STUPPIT:基于两步酶促反应的精准标记策略
研究团队通过巧妙整合split-TurboID和PUP-IT两种技术,开发了STUPPIT(Split-TurboID and PUP-IT based Protein Identification Tool)。其核心机制基于一个精确设计的“两步走”策略:
第一步,将PafA酶与诱饵蛋白A融合,将其底物PupE与TurboID的N端片段(TbN)融合。PafA酶可催化底物PupE-TbN与诱饵蛋白A邻近的蛋白(包括中间蛋白B)发生偶联。
第二步,当被PupE-TbN标记的中间蛋白B脱离诱饵蛋白A,转而与融合了TurboID C端片段(TbC)的蛋白C结合时,两个TurboID片段重新组装,恢复生物素连接酶活性,从而将生物素标记到中间蛋白B、蛋白C及其邻近蛋白上。这些带有生物素标记的蛋白随后可被链霉亲和素进行富集和鉴定。
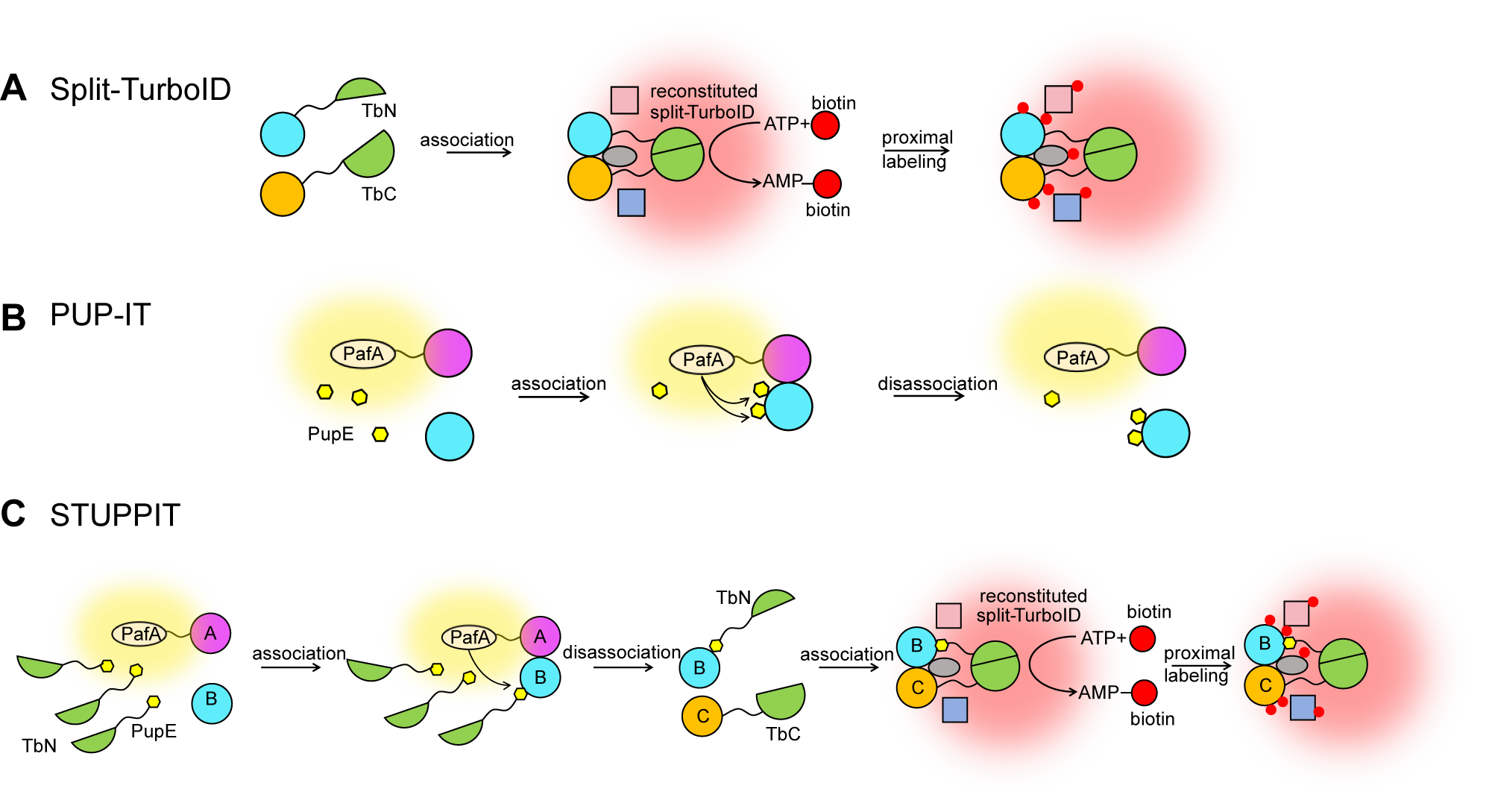
STUPPIT工作原理图
Ø 成功识别并验证新型“桥梁蛋白”
研究团队利用机械力信号转导中多对已知的蛋白质三元复合物(YAP1/AMOT/β-actin、YAP1/LATS1/MOB1A、β-catenin/α-catenin/β-actin)验证了STUPPIT的有效性,均成功捕获到已知的中间蛋白。
为进一步评估该技术的应用潜力,团队聚焦于Wnt及BMP信号通路。鉴于二者在毛囊干细胞和肠道干细胞命运决定中存在拮抗作用,推测其可能通过竞争结合某一中间蛋白实现相互拮抗。研究者将STUPPIT应用于Wnt和BMP信号通路的核心蛋白β-catenin和SMAD4,通过质谱分析发现ERC1和USP7等可能是二者的中间蛋白,后续通过免疫共沉淀及PUP-IT方法证实二者既能与β-catenin互作,也能与 SMAD4结合,提示它们在Wnt和BMP信号通路的相互作用中扮演重要角色。
Ø 打开信号网络研究新大门
STUPPIT工具实现了对非直接相互作用蛋白质之间 “桥梁”蛋白的直接、特异性标记和捕获。该技术不仅突破了领域内长期存在的技术瓶颈,还有望广泛应用于多种生命过程及疾病机制的研究,为揭示复杂细胞信号网络提供了强有力的工具。
云南大学生命科学学院的研究生谢淋、高丽娟和博士后付卫红为该论文的共同第一作者。宁文秀和李华为通讯作者。感谢云南大学武旭娜教授提供的质谱分析支持,云南大学生命科学中心提供的成像技术支持,以及云南大学付文祥、陈茂荣、孙建伟教授、上海科技大学庄敏教授和北京大学陈鹏教授提供的相关质粒。本研究得到国家自然科学基金No. 32270846, W.N和云南省基础研究计划面上项目基金No. 202401AT070443, H.L和云南大学人才启动经费的资助。
原文链接:https://doi.org/10.1371/journal.pbio.3003227
温馨提示:宁文秀课题组主要从事皮肤干细胞命运决定机制研究,欢迎感兴趣的同学、博士后加入课题组! 课题组链接:http://www.ynusky.ynu.edu.cn/info/1055/1902.htm
